 ¿QUÉ SON LOS ELEMENTOS MÓVILES?
¿QUÉ SON LOS ELEMENTOS MÓVILES?
Los elementos móviles son fragmentos de ADN que tienen la habilidad de saltar de una parte a otra del genoma. Mientras que la mayoría del ADN está siempre en el mismo lugar rodeado de los mismos trozos de ADN vecinos, los elementos móviles se mueven de un lugar a otro, pudiendo provocar muchos cambios o mutaciones. Podríamos decir que son como los rebeldes del genoma.
Algunos de estos cambios hacen que el organismo tenga más posibilidades de sobrevivir y/o reproducirse y, por tanto, más posibilidades de transmitir este cambio a su descendencia.
Un elemento móvil (o «elemento transponible» o «TE» por las siglas en inglés de transposable element) es un fragmento de ADN que puede cambiar su posición dentro del genoma, como si fuera un saltamontes, ya sea directamente cortándose y pegando en otro lugar (transposones) o indirectamente por acción de un ARN intermediario (retrotransposones). Están presentes en todos los organismos vivos que han sido estudiados, a excepción de algunos (como por ejemplo la bacteria Bacillus subtilis y los protozoos parasitarios Leishmania y Plasmodium falciparum).
Son más abundantes en eucariotas* que en procariotas*. Por ejemplo, en los humanos más de los dos tercios de su genoma son elementos móviles, en Drosophila melanogaster, un 10-15%, y en el maíz, un 80-85%. En los eucariotas, se puede dar un salto de un cromosoma a otro o un salto de un lugar a otro en un mismo cromosoma. En cambio, en los procariotas, saltan entre ADN cromosómico, entre ADN y plásmido* o entre dos plásmidos.
- * Eucariota: Organismos cuyas células presentan núcleo.
- * Plásmidos: Molécula de ADN circular propia de los procariotas.
- * Procariota: Organismos formados por una célula que no presenta membrana rodeando el material genético (es decir, sin núcleo).
¿CÓMO LOS PODEMOS CLASIFICAR?
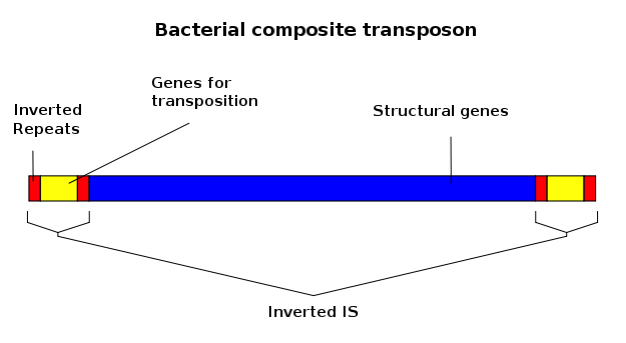
 Los elementos móviles se pueden clasificar en dos grandes grupos: los de clase I (retrotransposones) y los de clase II (transposones).
Los elementos móviles se pueden clasificar en dos grandes grupos: los de clase I (retrotransposones) y los de clase II (transposones).
Los transposones son aquellos que se desplazan directamente de un lugar a otro en el genoma utilizando una enzima (transposasa) para cortar y pegar en otro lugar. Dos ejemplos de este tipo son los activadores, que funcionan de manera independiente, y los disociadores, que dependen de los activadores. Ambos fueron estudiados por Barbara McClintock* en cepas de maíz.
Por otro lado están los retrotransposones. Al igual que en el caso de los transposones, la célula huésped reconoce el retrotransposón como una secuencia de ADN normal, y hace una copia de ARN. En el caso de los retrotransposones este ARN se convierte en ADN mediante una enzima llamada retrotranscriptasa. Los retrotransposones se subclasifican en dos tipos: víricos y no víricos. Dos ejemplos de retrotransposones víricos son los transposones Ty y los transposones copia. En cambio, entre los retrotransposones no víricos se encuentran ejemplos como los LINEs, que codifican transcriptasa inversa y son transcritos por ARN polimerasa II; los SINEs, que no codifican transcriptasa inversa y son transcritos por ARN polimerasa III, y los elementos Alu.
 Barbara McClintock (1902 – 1992) fue una científica estadounidense especializada en citogenética que fue premiada en solitario con el premio Nobel de Medicina o Fisiología en 1983, por sus estudios y descubrimientos sobre el maíz en los años cuarenta y cincuenta. McClintock descubrió el proceso de transposición de elementos del genoma y lo empleó para explicar cómo los genes determinan ciertas características físicas. Desarrolló hipótesis sobre la regulación de la expresión génica y la transmisión de los caracteres de plantas “madre” a plantas “hijas” de maíz. Además, por el gran nivel de su trabajo científico fue galardonada en varias ocasiones, por lo que entró a formar parte de la Academia Nacional de Ciencias de los Estados Unidos.
Barbara McClintock (1902 – 1992) fue una científica estadounidense especializada en citogenética que fue premiada en solitario con el premio Nobel de Medicina o Fisiología en 1983, por sus estudios y descubrimientos sobre el maíz en los años cuarenta y cincuenta. McClintock descubrió el proceso de transposición de elementos del genoma y lo empleó para explicar cómo los genes determinan ciertas características físicas. Desarrolló hipótesis sobre la regulación de la expresión génica y la transmisión de los caracteres de plantas “madre” a plantas “hijas” de maíz. Además, por el gran nivel de su trabajo científico fue galardonada en varias ocasiones, por lo que entró a formar parte de la Academia Nacional de Ciencias de los Estados Unidos.
¿CÓMO PUEDEN AFECTAR AL GENOMA?
Los elementos transponibles pueden provocar diferentes cambios o mutaciones dentro del genoma.
Para empezar, cuando un transposón se mueve por el genoma se puede insertar en un gen vital, perturbando su función, o en un lugar cerca de genes vitales, causando una activación errónea o una inactivación.
Por otro lado, puede haber reorganizaciones cromosómicas debido a transposones de la misma familia con secuencias complementarias que se encuentran separadas en el genoma y que por homología de secuencias emparejan. Esto puede dar lugar a la recombinación ectópica*. Las reorganizaciones a gran escala inducidas por elementos móviles se consideran una clase particular de eventos de recombinación que influyen en la plasticidad genómica; pueden provocar desde inversiones o duplicaciones hasta deleciones de hasta cien kilobases.
Una de las mayores contribuciones de los elementos transponibles a la evolución del genoma del huésped es proporcionar una fuente de material genético para crear nuevos genes y nuevas funciones. Los elementos transponibles tienen numerosas propiedades que los predisponen para ser integrados por el genoma del huésped para ganar ventajas funcionales.
- * Recombinación ectópica: Recombinación entre secuencias de cromátidas (brazo del cromosoma) no hermanas.
¿QUÉ EFECTOS PUEDEN PROVOCAR?
Estas modificaciones en el genoma pueden tener efectos positivos, negativos o neutros.
Existe un gran número de mutaciones que pueden ser perjudiciales y pueden causar enfermedades, como por ejemplo la hemofilia A y B, la neurofibromatosis o la hipercolesterolemia familiar, que están causadas por inserciones de elementos Alu o por la recombinación ectópica de diferentes copias de SINEs.
Por otro lado, un ejemplo de mutación beneficiosa es la que se da a los telómeros *, que consiste en repeticiones en tándem de dos retrotransposones HeT-A y TART. Su actividad transposicional compensa la ausencia de la enzima telomerasa. Esta se puede observar en Drosophila melanogaster.
Un ejemplo de mutación neutra se puede ver en las petunias, donde la línea genética W138 proviene de la línea salvaje R27. Su principal diferencia es que una mutación en el locus AN1, encargado de la producción de los pigmentos de la flor, produce una coloración irregular.
- * Telómero: extremo del cromosoma.
¿CÓMO PUEDE SER QUE, TENIENDO TANTOS ELEMENTOS MÓVILES QUE PUEDEN CAUSAR TANTAS MUTACIONES, SIGAMOS VIVOS?
Existen diferentes mecanismos que regulan y controlan los elementos móviles. Estos mecanismos son comunes en algunos organismos y específicos en otros. Sin embargo, siempre están presentes.
Existen mecanismos de control generales de los genomas eucariotas, que consisten en el silenciamiento génico y actúan a diferentes niveles. En primer lugar, el silenciamiento transcripcional actúa sobre las regiones promotoras de los genes, bloqueando la transcripción. El silenciamiento post-transcripcional actúa a nivel de ARN, degradándose o bloqueando su traducción.
 Un ejemplo de silenciamiento génico que encontramos en la Drosophila melanogaster, entre otros, son los ARN asociados a Piwi (piRNAs). Los piRNAs se generan a partir de regiones del genoma donde hay diferentes elementos móviles insertados. Cuando se transcribe esta región se forma un transcrito que contiene diferentes copias de los elementos móviles. Este ARN se corta en trozos pequeños, de 24-31 nucleótidos, llamados piRNAs. Estos piRNAs se unen a una proteína del grupo Piwi, que conduce al piRNA hasta la secuencia complementaria de ARNm (ARN mensajero) del elemento móvil de la misma familia. Una enzima asociada a Piwi degrada los ARNm del TE y libera una cadena de piRNA, que se puede asociar con otras proteínas Piwi. La unión de este complejo en el transcrito inicial que contiene diferentes copias de los elementos móviles regenera un piRNA complementario al anterior que puede ser dirigido al ARNm del TE. Este ciclo se conoce como «ciclo ping-pong».
Un ejemplo de silenciamiento génico que encontramos en la Drosophila melanogaster, entre otros, son los ARN asociados a Piwi (piRNAs). Los piRNAs se generan a partir de regiones del genoma donde hay diferentes elementos móviles insertados. Cuando se transcribe esta región se forma un transcrito que contiene diferentes copias de los elementos móviles. Este ARN se corta en trozos pequeños, de 24-31 nucleótidos, llamados piRNAs. Estos piRNAs se unen a una proteína del grupo Piwi, que conduce al piRNA hasta la secuencia complementaria de ARNm (ARN mensajero) del elemento móvil de la misma familia. Una enzima asociada a Piwi degrada los ARNm del TE y libera una cadena de piRNA, que se puede asociar con otras proteínas Piwi. La unión de este complejo en el transcrito inicial que contiene diferentes copias de los elementos móviles regenera un piRNA complementario al anterior que puede ser dirigido al ARNm del TE. Este ciclo se conoce como «ciclo ping-pong».
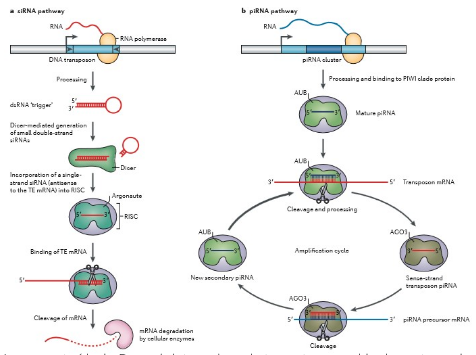
Por otra parte, también existen otros mecanismos presentes en humanos como pueden ser los siRNA (small interfering RNA) o ARN de interferencia (ARNi). Estos, al igual que los piRNAs funcionan por homología de secuencias. En primer lugar, se forma el transcrito como en el caso de los piRNAs. Este transcrito tiene la peculiaridad de que está formado por una secuencia con terminaciones largas invertidas, las cuales son complementarias entre sí y, por tanto, se aparean formando una estructura de ARN con forma de horquilla, con doble cadena. La célula no reconoce el ARN doble como propio, por lo que mediante una proteína (Dicer), elimina el bucle. Una vez hecho esto, una de las dos cadenas se degradada y la otra cadena se une a otra proteína (Argonauta). En este punto, se forma el complejo llamado RISC. La cadena se empareja por homología de secuencias a un ARNm, para que, posteriormente, sea degradada por una enzima.
El ARN de interferencia puede ser iniciado mediante la exposición de las células a ARN ya sea a través de la transfección* o expresión endógena, es decir, dentro de la célula. En sistemas de mamíferos, el efecto del ARNi específico de secuencia se ha observado por la expresión de transcritos de 21-23 bases capaz de formar dúplex, o a través de la expresión de ARN de horquilla corta.
Otro mecanismo es la metilación del ADN, que es la adición de un grupo metilo a los nucleótidos de citosina. Hay dos hipótesis sobre cómo la metilación puede afectar a la transcripción de los genes. En primer lugar, la metilación del ADN en sí puede impedir físicamente la unión de las proteínas necesarias para la transcripción del gen, interponiéndose entre ambos. Para realizar la transcripción, la cromatina debe estar descompactada para que se pueda leer la información génica. La segunda hipótesis dice que se añaden proteínas al ADN metilado, compactando la cromatina y dando lugar a heterocromatina. Esta unión da lugar a un cambio de estructura que provoca que no se pueda transcribir el gen.
En conclusión, el control de la transposición es fundamental para la supervivencia de los diferentes organismos. Sin embargo, este control no implica la inactivación definitiva de los efectos de ciertos elementos móviles.
- * Transfección: introducción de material genético externo en células mediante plásmidos o vectores víricos.
Redacción: Henry Estrella y Mar Gutiérrez, estudiantes del Programa “Jóvenes y Ciencia” que realizaron su estancia en González Lab



